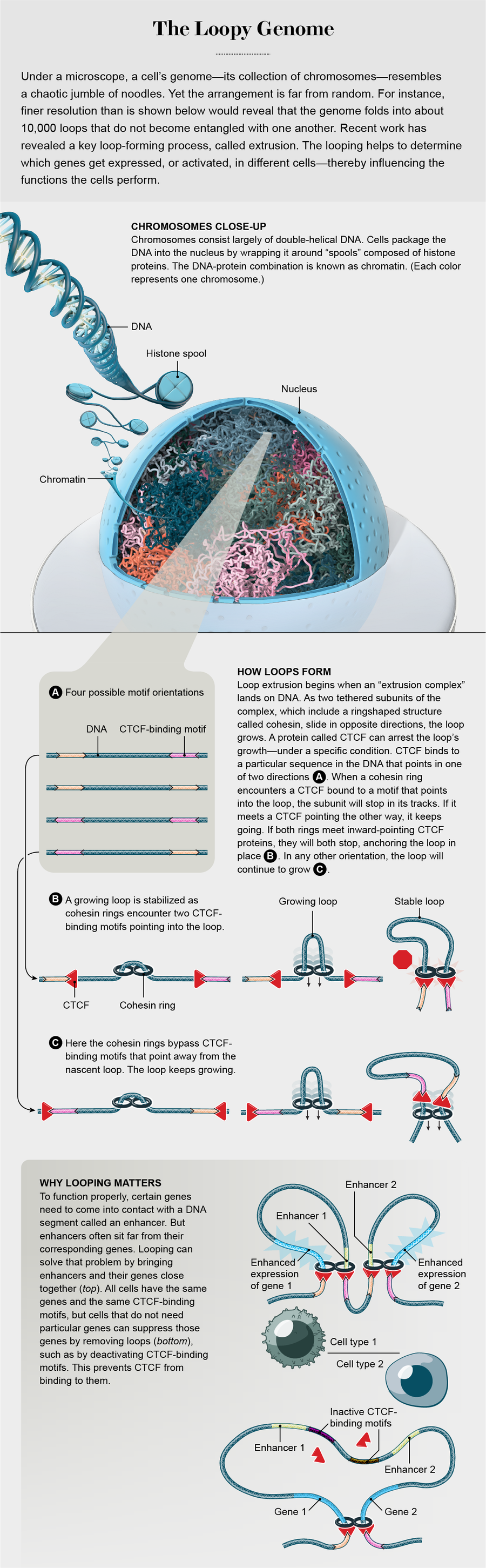
因为我发现很难将人类基因组的结构这样微小的东西联系起来,所以我喜欢把它放大一百万倍来想象。在这个尺寸下,每个DNA分子——一条染色体——都像拉面条一样宽。如果将构成细胞基因组的全部46条放大后的染色体首尾相连,它们将从纽约延伸到堪萨斯城,尽管它们实际上是折叠起来以适应房屋大小的结构——细胞核内部。总共有46条染色体,包含大约两组20,000个基因。每个基因都拼写出一个编码信息,告诉细胞如何制造特定的蛋白质;在百万倍的尺度下,一个基因和一辆汽车一样长。
窥视细胞核内部,你会看到DNA在不停地摆动。大约十年前,当我还是博士生的时候,我一边搅拌晚餐里的拉面条,一边想知道基因组是如何避免像我的面条一样缠结成一团糟,从而阻止其关键的遗传信息被发送出去的。
2014年,我和我的同事为这个问题的一个答案做出了贡献,进一步认识到细胞核内部基因组的结构远非随机。我们位于贝勒医学院的团队,由我的学生Suhas Rao、Miriam Huntley和Adrian Sanborn领导,发现人类基因组以形成约10,000个环的方式折叠。这些环遵循一个简单的代码,隐藏在基因组本身的序列中。事实证明,它们是古老的结构;许多相同的环也出现在小鼠中,这是6000多万年前的祖先物种的共同遗产。这种跨越时间的持久性表明,这些环对生存至关重要。
支持科学新闻报道
如果您喜欢这篇文章,请考虑通过以下方式支持我们屡获殊荣的新闻报道 订阅。通过购买订阅,您正在帮助确保有关塑造我们当今世界的发现和想法的具有影响力的故事的未来。
这些环似乎有助于控制基因活动。所有细胞都具有相同的基因,但是如果活动模式没有差异,身体就不可能存在:心肌细胞与脑细胞将没有区别。这些独特的模式是如何协调的,一直是一个谜。现在看来,环是模式控制器之一,是基因管弦乐队的指挥,影响着特定基因何时变得足够活跃以影响细胞功能。
随着我们继续探索这些环,我们期望更好地理解基因调控,并找到许多疾病如何产生的线索。最近,我们和其他人已经弄清楚了这些环是如何形成的,跳着优雅的探戈,使基因组保持无缠结状态。
基因组的Facebook
我对DNA缠结的思考与一个更大的问题有关:细胞核中DNA的3-D排列如何影响基因活动?自1970年代后期以来,越来越多的证据表明,激活基因需要称为增强子的DNA小片段。生物学家还了解到,增强子可以位于DNA链中远离其靶基因的位置。为了触发基因的“开启”开关——基因附近的DNA片段,称为启动子——该链大概必须自身环回,使增强子靠近启动子。但是这种假设正确吗?我被这个问题迷住了,只能想到一种解决它的方法:找到所有环。
从概念上讲,这样做的计划很简单。如果两个人特别频繁地在一起,那么可以逻辑地推断他们是朋友。同样,我们推断,如果染色体上相距遥远的两段DNA(“位点”)倾向于特别频繁地在一起,那么DNA可能已经折叠成一个环。我们需要的是一种方法来测量基因组的各个部分相互作用的频率:为人类基因组构建类似Facebook的东西。
为了将我们的想法变为现实,我们采用了1993年当时在范德比尔特大学的Katherine Cullen及其同事描述的一种方法。当时,基因组使所有已知的成像形式都感到困惑:就像一个糟糕的肖像对象一样,摆动的染色体面条拒绝静止不动。但是,库伦利用了这种不稳定的染色体来发挥她的优势。她知道,随着染色体的晃动,基因组的不同部分会相互碰撞。在3-D中非常接近的部分会相互碰撞很多次;相距遥远的部分只会偶尔接触。因此,如果您可以测量碰撞频率,则可以弄清楚基因组的哪些部分在3-D空间中彼此靠近。
为了测量这种碰撞频率,库伦和她的同事开发了他们称之为核连接测定(NLA)的方法。本质上,您取出细胞,并在不破坏其细胞核的情况下,稳定其基因组。然后,您送入一种酶将DNA切成小片段,并部署一种蛋白质,将两个附近片段的末端融合,形成单链。最后,您检查融合片段集合中DNA碱基对(DNA代码的配对字母,形成DNA熟悉的“梯子”的“梯级”)的序列。如果在细胞接细胞中,您看到特定DNA片段对的融合,而这些片段最初并非在染色体上彼此相邻(称为连接连接点),则可以得出结论,这两个DNA片段经常在细胞核的3-D空间中彼此靠近。
库伦在科学杂志上发表的见解使她能够证明,DNA特定长片段两端的两个DNA片段比偶然预测的频率更高地相互碰撞。换句话说,DNA形成了一个环。
早在1993年,使用核连接测定进行的实验很难执行。幸运的是,当我在2000年代中期作为研究生看到库伦的论文时,已经有了一个可用的参考人类基因组,并且DNA测序变得非常便宜。我和麻省理工学院和哈佛大学布罗德研究所的其他三个人——Chad Nusbaum、Andreas Gnirke和Eric Lander——勾勒出一种方法,该方法将分析的接触频率不是单个DNA位置对的接触频率,而是整个基因组中每个位置对的接触频率。它还将使我们能够精确地查明每个连接连接点的一半来自何处。
我们决定将我们的新方法建立在马萨诸塞大学医学院的Job Dekker开发的库伦程序变体之上。与库伦使用完整细胞核不同,德克尔炸开了细胞核,并在极其稀释的溶液中执行了关键的连接步骤。德克尔普及并称为“染色体构象捕获”或“3C”的这种修改,被认为可以产生更可靠的碰撞频率估计值。
接下来,我们在3C中添加了一些步骤。在将片段粘合在一起之前,我们将在破碎的DNA末端附上易于检测的标签——以标记两个附近片段连接的位置。完成此步骤后,我们将把粘合的片段切成更小的片段,并仅拉出带有标签的片段;这些片段将包含纯连接连接点。与德克尔、他当时的博士后研究员Nynke van Berkum和布罗德研究所的Louise Williams合作,我们发现我们可以一次识别数百万个接触。我将该方法称为“Hi-C”,这是对“3C”和我小时候最喜欢的饮料之一的名称的模仿。我们在2009年发表了该方法。
我们最早的全基因组Hi-C图谱显示,染色体尽管不停地摆动,但并没有在细胞核内随机混乱地折叠起来。相反,每条染色体都被划分为域:包含彼此频繁接触的片段的DNA片段。一个域中的位点与另一个域中的位点相互作用的频率较低。更重要的是,我们的Hi-C数据揭示,每个域都位于细胞核中两个较大的空间邻域之一内。我们将这些邻域称为“区室”,并标记为A和B。
我们发现A区室富含遗传活动标记,例如信使RNA,信使RNA是基因发送出去以告知细胞其余部分该做什么的分子。B区室更密集,并且基本上处于非活动状态。当域开启或关闭时,它们会从一个区室移动到另一个区室。(今天我们知道细胞核包含多个A和B亚区室。)
这种动态区室化的发现使我们感到兴奋,因为它证实了基因组的大规模3-D结构并非随机的,而是与基因活动密切相关的。但是,我感到失望的是,Hi-C数据中似乎从未出现过一种折叠特征:环!
Hi-C数据通常表示为热图:一个图,显示染色体中两个位点相互形成接触的频率。在这样的图中,两个位点之间的接触频率由x轴和y轴上点的亮度表示,代表位点的交点。环应该表现为一个异常明亮的点,对应于环的两个锚点。但是我们没有看到任何这样的亮度峰值。如果我们不能证明环正在形成,我们就无法探索增强子是否通过物理上靠近启动子来激活基因。
制作环图
这个问题困扰了我们三年。然后,在2012年,Rao和Huntley弄清楚了哪里出了问题。他们意识到Hi-C的一个方面——在连接之前破坏细胞核——破坏了环等精细结构。因此,他们着手开发一种更新的Hi-C方法,该方法在连接过程中保持细胞核完整。
这种称为原位Hi-C的新方法产生了巨大的不同。在对白细胞的研究中,Rao和Huntley发现,明亮的峰值现在出现在我们热图的各个位置,每个峰值代表一个推定的环。但是,自从我开始致力于绘制环图以来,已经过去了六年;我不再相信自己的眼睛。我的团队和我担心我们可能在数据中看到了实际上不存在的东西。
为了确保我没有受到确认偏差的影响,我把地图带回家给我的儿子加布里埃尔,当时他三岁。“你看到一个红点吗?”“是的,”他说。“你能指出来吗?”他可以。
我们成功了:一张显示10,000个环的地图,分布在整个人类基因组中。我们检查了这些环是否连接了基因启动子和增强子。它们经常这样做。
在进一步的测试中,我们将我们的血细胞图谱与另一种细胞——来自肺部的细胞的新图谱进行了比较。我们看到了许多相同的环,但我们也看到了我们推测涉及不同增强子和不同靶基因的新连接。环模式的这些变化表明,环可能参与调节赋予细胞独特身份的基因。
我们想知道环化是否是人类独有的,或者其他生物体中是否存在相同的环。因此,我们绘制了小鼠细胞中环的图谱,发现一半的环存在于人类基因组的相应位置。这些共享的环在至少6000万年的进化过程中得以保存,从科罗拉多河开始雕刻大峡谷之前很久就在地球上漫游的祖先生物开始。
环的逻辑
我们数据的一个有趣的含义是,环不是静态的:它们似乎不断产生、分离并再次形成。自然,我们想知道这是如何工作的。
我们怀疑数百种蛋白质参与其中。然而,数据讲述了一个不同的故事。在一个又一个的环中,突出了两个蛋白质因子。一个名为CTCF,由Victor Lobanenkov及其同事于1990年发现。它包含11个称为锌指的成分,使CTCF能够非常紧密地结合到DNA上的特定位点。第二个因子,粘连蛋白,由现任牛津大学的Kim Nasmyth于1997年发现,是一个由多种蛋白质组成的环状复合物。人们认为,两个粘连蛋白环可能会连接起来并协同工作,这对环中的每个环都环绕DNA并在其上自由滑动,就像项链上的环一样。
图片来源:Falconieri Visuals
看到这些蛋白质并不完全令人惊讶:许多早期的研究表明它们可能参与基因组折叠,尽管在环锚点(尤其是在连接启动子和增强子的环处)起如此普遍的作用是出乎意料的。
然后我们偶然发现了一些真正奇怪的东西。Rao、Huntley和我要求实验室的一位新的计算科学家Ido Machol研究组蛋白(有助于将DNA包装在细胞核内部)在CTCF分子附近的分布。Machol注意到,环外侧的组蛋白比内侧的组蛋白更多,就好像组蛋白以某种方式知道环相对于CTCF分子的位置一样。我怀疑这个发现只是反映了Machol代码中的一个错误。但是随着时间的推移,Machol没有发现任何错误。
我们开始寻找生物学解释。在描述CTCF发现的原始论文中,Lobanenkov表明CTCF并非附着在DNA上的任意位置。相反,它总是结合到特定的DNA词——大约20个碱基的特定序列,称为基序。由于DNA是双螺旋结构,因此它具有两条链。基序可以出现在任一条链上,指向巨大的DNA面条的任一端。DNA基序的相对方向通常是随机的,就像抛硬币一样:典型的基序有50%的几率指向一个末端,而有50%的几率指向另一个末端。因此,起初,我们希望看到环锚点处CTCF结合基序的随机方向。
我们想知道环锚点处的CTCF结合基序是否给组蛋白提供了线索,告诉它们应该在哪里连接到基序附近的DNA。我们进行了检查,令我们惊讶的是,即使两个微小的CTCF结合基序在未折叠的DNA中相隔数百万个DNA字母,它们也总是彼此指向,并指向环内,我们将其命名为会聚方向。这种会聚规则解释了组蛋白如何知道自身定位的位置——它们只需要确定CTCF结合基序的指向方向即可。
但是,在解决一个难题的同时,会聚规则又制造了第二个更大的谜团。基序的非随机方向与预期相反。为了便于理解,让我们再次将基因组放大一百万倍。现在,基序的长度均为五毫米,并且被长达一公里的基因组面条分隔开。然而,不知何故,仿佛受到神奇指南针的引导,形成环状DNA片段两端的基序总是彼此指向。像任何好的魔术技巧一样,会聚规则似乎在物理上是不可能的。这也与人们普遍接受的环可能形成的方式相矛盾。
当时,几乎所有人(包括我们自己)都认为基因组环是通过扩散形成的。在该方案中,形成环所需的蛋白质在DNA片段的一端结合。接下来,另一个环使能蛋白在另一端结合。然后,像往常一样,DNA摆动。最后,如果摆动使两种蛋白质结合在一起,它们就会形成物理连接,从而形成一个环。问题是,整个DNA链有太多的摆动空间,如果扩散模型是正确的,那么CTCF结合基序的相对方向就无关紧要了。然而,我们却看到了会聚。在一年之内,由伦敦大学学院的Suzana Hadjur领导的一个团队和由杰克逊实验室的Yijun Ruan领导的一个团队在他们自己的数据集证实了会聚规则。该规则将继续存在,我们看到的环不可能通过扩散形成。
驱动式环化
如果环不是通过扩散形成的,那么它们是如何产生的?CTCF和粘连蛋白的作用是什么?在基因组折叠紧急情况下,我们总是会这样做:我们开始摆弄耳机线。
我非常确定,大多数从事基因组折叠工作的人都会随身携带一个长长的面条状物体:一根纱线,一根塑料管。当您遇到难题时,您会拿出这个物体并摆弄一番。有一天,当我们探索环形成的可能模型时,Rao和我来回传递耳机。突然,我们意识到答案不在我们的耳机中;而是在我们的背包上。
想象一下调节背包带长度的装置。这个物体称为三滑扣,或多或少由两个彼此物理连接的环组成。带子从第一个环进入,然后从第二个环出来。如果您想调整带子长度,则可以将一些带子拉过其中一个环并开始形成一个环。您可以继续扩大环的尺寸,直到到达一块折叠的材料阻止您。
也许成对的粘连蛋白环像三滑扣一样工作?起初,它们附着在基因组上的任何位置,DNA进入一个环并从另一个环出来。但是随后,这两个环沿相反的方向滑动(一个沿线性分子向左滑动,一个向右滑动),在滑动过程中挤出一个不断增长的环。但是它们不会永远滑动。最终,一个环接近CTCF分子结合的位点。如果底层的CTCF结合基序指向接近的环,则滑动环会在接触时停止。但是,如果基序朝另一个方向,则粘连蛋白会忽略它并继续前进。(通过这种方式,CTCF结合基序就像粘连蛋白交通的停车标志:如果标志朝向您,则您停止;如果标志朝向另一个方向,则您不会停止。)第二个环继续前进,直到它也到达一个朝内指向的CTCF结合基序。现在环已完成。
如果粘连蛋白环实际上以这种方式工作,那么环将仅在符合会聚规则的CTCF结合基序对之间形成。我们很快意识到,这种挤出过程将为细胞提供至关重要的好处。如果环通过扩散形成,那么染色体中的环对很容易缠绕在一起,导致染色体形成结并相互缠结。这将使基因难以正常运作,并可能阻止染色体在细胞需要分裂时分离。相反,通过挤出产生的环不会形成结或缠结——这就是为什么无论您用三滑扣调整背包带的长度多少,背包带都不会打结的原因。
该模型是疯狂的猜测。它做出了许多我们没有直接证据的基本假设,例如粘连蛋白可以沿DNA滑动的概念。我们担心我们疯了。但是,当我们阅读有关粘连蛋白的文献时,我们意识到Nasmyth本人早在2001年就提出粘连蛋白可能会挤出DNA。Sanborn运行了详细的模拟,这些模拟密切概括了我们地图中的数据。当Rao在真实DNA上进行实验时,环化变化的方式与Sanborn的模型预测的完全相同。
删除环锚点处的CTCF结合基序消除了环。翻转基序的方向会使原始环消失,但会在另一侧形成另一个环。添加CTCF结合基序——只要它指向正确的方向——也会导致新环的形成。然后我们发现我们可以随意地向基因组添加和删除环。
我们迅速撰写并提交了一篇关于我们的挤出模型和我们为测试该模型而进行的环工程实验的论文。该领域正在升温,在2015年末的几个星期内,我们的实验室和其他两个团队发表了论文,证明了这种3-D基因组手术是有效的。同样,三个团队——我们的团队、埃默里大学的一个团队和麻省理工学院的一个团队——报告说,会聚规则支持环通过挤出形成的模型。最终,科学界开始解开环的逻辑。
进展仍在继续,现在正以惊人的速度进行。在格莱斯顿研究所,Benoit Bruneau及其同事表明,干扰CTCF会大大削弱环。在欧洲分子生物学实验室,Francois Spitz及其同事通过消除一种被认为将粘连蛋白加载到DNA上的蛋白质而获得了类似的结果。在荷兰癌症研究所,Benjamin Rowland的团队表明,消除一种从DNA中去除粘连蛋白的因子会导致更大的环,这可能是因为粘连蛋白现在可以滑动更长的时间。在我们实验室,Rao表明,通过降解粘连蛋白本身,我们可以在几分钟内消除所有粘连蛋白环。
但是我们都渴望得到直接的证实:亲眼目睹挤出作用。最终,在2018年4月,荷兰代尔夫特理工大学的Cees Dekker及其同事做到了这一点。通过使用酵母的缩合蛋白——一种与粘连蛋白密切相关的蛋白质复合物——他们制作了一部显微电影,核结构领域的许多人将永远不会忘记。首先,您会看到一条DNA带。然后,缩合蛋白着陆,形成一个小的DNA结节。结节不断生长,直到观看者意识到它的真实含义:一个挤出的环。
转向健康
随着环形成的机制和规则的出现,环化对于健康和疾病的重要性变得越来越清晰。例如,哈佛大学的Frederick Alt及其同事已经开始阐明环化在抗体产生中起的作用。您的身体通过剪切和粘贴抗体基因片段来制造以前从未遇到过的病原体的抗体。Alt的团队发现,这个过程是通过形成多个CTCF锚定的环,然后将它们切除来完成的。
柏林马克斯·普朗克分子遗传学研究所的Stefan Mundlos实验室表明,修饰小鼠中的单个CTCF结合基序会导致动物的爪子发育出异常数量的脚趾。具有相应变化的人类没有五根手指。美国国立卫生研究院的Rafael Casellas表明,破坏小鼠浆细胞瘤(一种癌症)中的CTCF结合基序可以使肿瘤的生长速度减慢40%。
然而,随着环挤出概念越来越受到重视,关于环在基因调控中起作用的更深层理论开始瓦解。几十年来,科学家们认为环像开关一样工作:当增强子和启动子之间的环存在时,相应的基因就会开启。因此,我们预计,当我们从细胞中去除粘连蛋白时,基因表达会变得混乱,成千上万的基因会改变其活性水平。正如预测的那样,许多基因确实发生了变化。但是变化相当小。环——至少是挤出形成的环——毕竟不是二元开关。相反,它们似乎更像旋钮,将基因活性向上或向下稍微调整一点,微调细胞的不同蛋白质的供应。
换句话说,大自然让我们陷入了一个循环。我们以为我们理解了游戏规则,环会开启基因。但是现在我们已经看到了环的实际作用,我们必须承认我们的视野过于简单了。甚至有可能基因调控可能只是环的副业;也许它们在细胞中的主要功能完全是另一回事。
像任何在未开发领域探险的探险家一样,我们需要更好的地图。我在美国国立卫生研究院的DNA元件百科全书(ENCODE)项目的同事Ruan和我目前正在与我们的同事合作,创建人类基因组中环化的第一个图谱,绘制整个人体组织中的环。我们的团队和许多其他团队也加入了4D核组联盟,该联盟正在开发解决这些问题的新方法。我的实验室的博士后Olga Dudchenko创建了DNA动物园——一个由世界各地的学术实验室、动物园和水族馆组成的联盟,该联盟正试图组装数百个物种的基因组,记录生命之树中环的进化。
对于研究人员来说,一个科学故事的结尾始终是另一个科学故事的开始。二十亿年前,在细胞核出现之前,DNA挤出过程就出现了。为什么?再次,进入循环。

